複数の仮想環境をJupyterのカーネルできるようにする方法
・Pythonの標準モジュールvenvを使って仮想環境(vm_1_venv※名前は適当)を作成する。
vagrant@ubuntu1804:~$ python3.7 -m venv vm_1_venv
・仮想環境を起動する。
vagrant@ubuntu1804:~$ . vm_1_venv/bin/activate
・Jupyterにカーネルを追加する。
(vm_1_venv) vagrant@ubuntu1804:~$ ipython kernel install --user --name=vm_1_venv --display-name=vm_1_venv
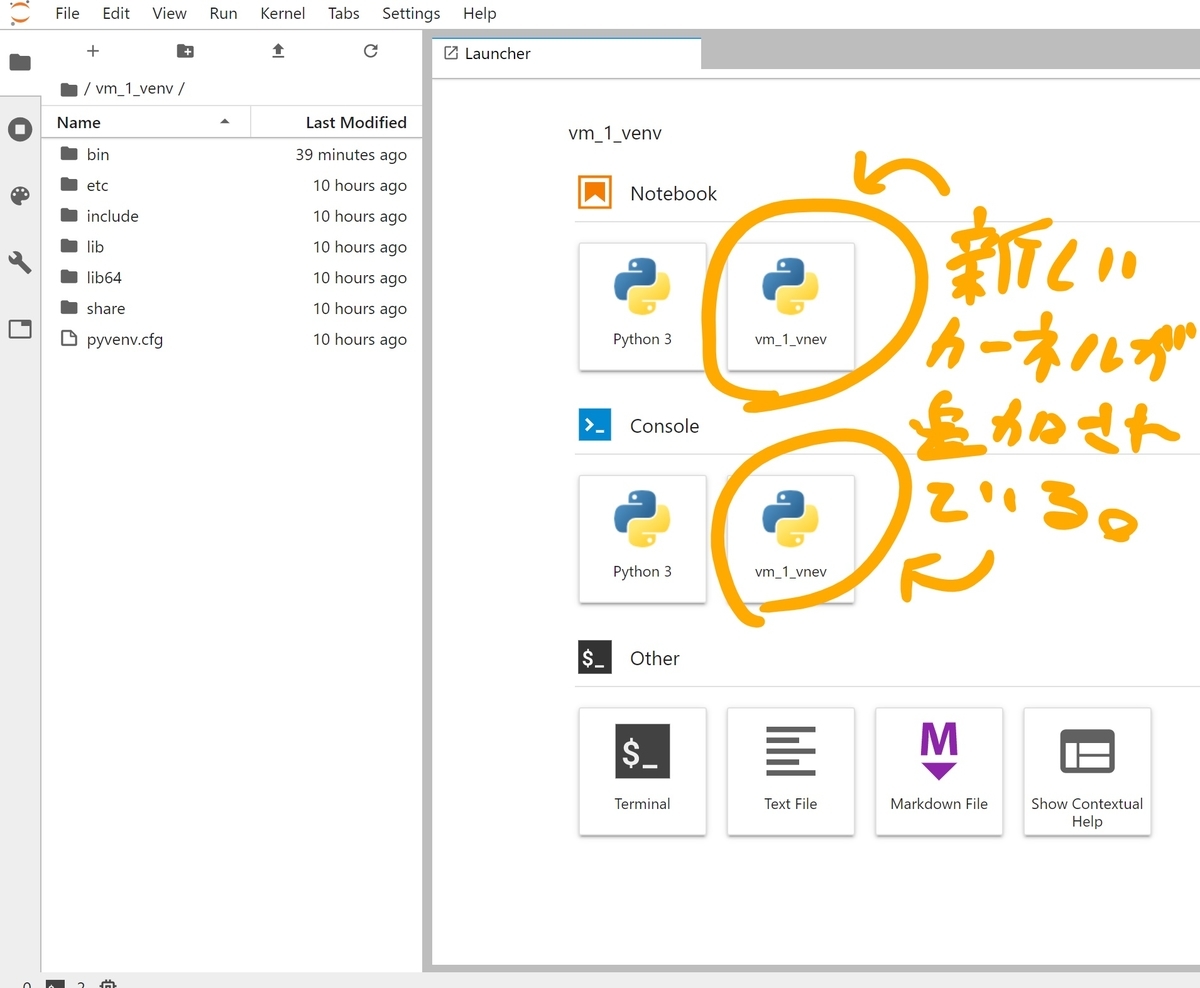
・以下のコマンドでカーネルが追加されたか確認する。元々のカーネルPython3と新しく追加したカーネルvm_1_venvが表示される。
(vm_1_venv) vagrant@ubuntu1804:~$ jupyter kernelspec list Available kernels: python3 /home/vagrant/.local/share/jupyter/kernels/python3 vm_1_venv /home/vagrant/.local/share/jupyter/kernels/vm_1_venv
・Jupyter NotebookまたはJupyter Labを起動して新しいカーネル(vm_1_venv)が追加されているか確認する。
venvで作成した仮想環境とAnacondaでインストールしたIPython(Jupyter)との関係
はじめに
venvで作成した仮想環境を使って、Jupyterで作業をしていたら、インストールしていないパッケージも使えたので、その原因を調べた。
venvで作成した仮想環境を通常のPythonのコンソールで使用する際の挙動
venvで作成した仮想環境をアクティベートした状態でターミナルから開いた通常のPythonのコンソール上では、仮想環境にインストールしていないパッケージを読み込むことはできない。
venvで作成した仮想環境をAnacondaを使ってインストールしたJupyterやIPythonで使用する際の挙動
venvで作成した仮想環境をアクティベートした状態で、IPythonやJupyterを使用すると、仮想環境にインストールしているパッケージは仮想環境のフォルダから読み込まれる。
仮想環境にインストールされていないパッケージは、仮想環境が乗っている本体のOSのAnacondaフォルダのPythonのパッケージフォルダから読み込まれる。
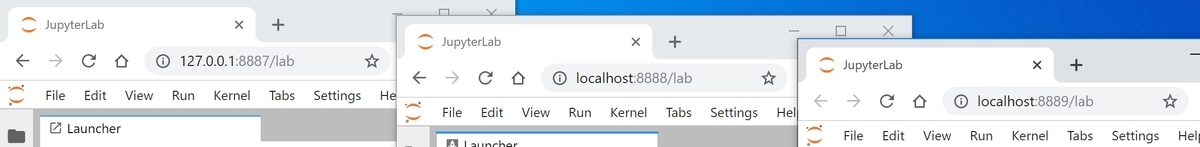
Jupyterを3つの環境(ホストOS、WSL、Vagrant)で同時に起動する。
はじめに
ホストOS(Windows)、WSL、VagrantにそれぞれJupyterをインストールしていて、それらを同時に起動して作業したい時に、上手く起動できない問題の解決策を紹介。結論としては、デフォルトのままだと、全てがローカルポート8888を使おうとしてエラーが生じるので、ローカルポートをうまく割り当てると解決する。
まずWSLで起動
C:\Users\*WinUSERNAME>bash (base) *LinuxUSERNAME@DESKTOP-XXXXXXX:/mnt/c/Users/*WinUSERNAME$ (base) *LinuxUSERNAME@DESKTOP-XXXXXXX:/mnt/c/Users/*WinUSERNAME$ cd ~ (base) *LinuxUSERNAME@DESKTOP-XXXXXXX:~$ jupyter lab
jupyter lab入力後に表示されるURLをブラウザに張り付けると、WSL上のJupyterが起動する。
※この時のローカルポートは8888である。
次にVagrantで起動
C:\Users\*WinUSERNAME\*vagrantのフォルダ>vagrant up C:\Users\*WinUSERNAME\*vagrantのフォルダ>vagrant ssh -- -L 8887:localhost:8888 (base) vagrant@ubuntu-bionic:~$ jupyter lab
ポイントは「vagrant ssh -- -L 8887:localhost:8888」とすること。
jupyter lab入力後に表示されるURLは「http://127.0.0.1:8888/?token=*xxxxxxxxxxxxxxxxxxxxxxxx」となってるので、この8888を8887にして(つまりhttp://127.0.0.1:8887/?token=*xxxxxxxxxxxxxxxxxxxxxxxx として)ブラウザに張り付けるとvagrant上のJupyterが起動する。
※この時のローカルポートは8887である。
最後にホストOS(Windows)で起動
Anaconda等からJupyter Labを起動すれば、自動的にローカルポートは8889(つまり8887と8888以外)に設定される。
Bash on Windows (Ubuntu OS)(WSL)でrootfsディレクトリに入れない原因と解決策
1.WSLでJupyter Labを開こうとするエラーが発生したので、確認のため、WSL上でUbuntuOSにインストールしたAnacondaのある場所まで進もうとすると、rootfsフォルダの中に入れない現象が発生し、非常に悩んだ。
(base) root@DESKTOP-XXXXXXX:/mnt/c/Users/*WINDOWS_USERNAME/AppData/Local/Packages/CanonicalGroupLimited.UbuntuonWindows_79rhkp1fndgsc/LocalState$ cd rootfs bash: cd: rootfs: Permission denied
rootfsのパーミッションの状況を確認するとオーナーなのに入れない謎の状況であることが判明。
(base) root@DESKTOP-XXXXXXX:/mnt/c/Users/*WINDOWS_USERNAME/AppData/Local/Packages/CanonicalGroupLimited.UbuntuonWindows_79rhkp1fndgsc/LocalState$ ls -l total 0 -rwxrwxrwx 1 tropicbird tropicbird 0 Oct 28 18:47 fsserver d--------- 1 tropicbird tropicbird 4096 Jun 30 08:35 rootfs drwxrwxrwx 1 tropicbird tropicbird 4096 Oct 28 18:47 temp
解決策を調べた結果、rootfsディレクトリ経由だとフォルダ検索に無限ループが発生することが原因であることが分かった。解決策はrootfsフォルダを経由せずにUbuntuOSのユーザーディレクトリに移動すればよいらしい。
※参考サイト↓
cannot access 'rootfs' on Ubuntu on Windows - Super User
3.WSL上でrootfs以降に進めない原因の解決策
コマンドプロンプトでbashを開いた時に最初のディレクトリの場所は以下の場所になっている。
(base) tropicbird@DESKTOP-XXXXXXX:/mnt/c/Users/*WINDOWS_USERNAME$
このままだと、無限ループになる可能性があるので、LINUXコマンドのcd .. を使ってディレクトリの一番上まで移動する。
(base) tropicbird@DESKTOP-XXXXXXX:/$
ここで、cd home/*LINUX_USERNAMEを入力すると、ターミナルの表示が少し変化する。
(追記2019/11/2:cd ..でフォルダの一番上まで移動してcd home/*LINUX_USERNAMEを入力しなくても、どこからでもcd~を入力すれば、一発で仮想環境のhomeに一発で移動できる。)
(base) tropicbird@DESKTOP-XXXXXXX:~$
これで、rootfsディレクトリによるループ現象を発生させずに、UbuntuOS上でインストールしたソフトやフォルダにエラーなく、アクセスすることができる。
ちなみに、lsを使って当方のhome/*LINUX_USERNAME内を確認してみると、以下のような感じ。
(base) tropicbird@DESKTOP-XXXXXXX:~$ls anaconda3 seaborn-data
4.cd home/*LINUX_USERNAMEからだとJupyter Labも問題なく開ける。
jupyter labと入力して、出てきたURLをブラウザで開くとエラーなく起動する。
(base) tropicbird@DESKTOP-XXXXXXX:~$ jupyter lab
WSLからAnacondaが開けない。
Bash on Windowを起動して、Anacoda Navigatorを起動してみるが、、
(base) tropicbird@DESKTOP-SL2TGNL:/mnt/c/Users/USERNAME$ anaconda-navigator
以下のようなエラーが発生する。
This application failed to start because it could not find or load the Qt platform plugin "xcb" in "". Available platform plugins are: eglfs, minimal, minimalegl, offscreen, vnc, xcb. Reinstalling the application may fix this problem. Aborted (core dumped)
解決策募集中です。
Bash on Windows のインストール方法
WindowsにBash(UbuntuOS)をインストールすることのメリット
・Windows上で普通にIPythonを開いて、「!UNIX系シェルコマンド」を実行してもうまくいかなかった。理由は通常のIPythonはWindowsOS上で動いているから。
・そのため、windowsの場合、IPython上でUNIX系のシェルコマンドを利用するには、次のステップが必要。つまり、UbuntuOS上にIPythonをインストールするということ。
1.Bash on Ubuntu on Windowsをインストール(上記のリンクの作業のこと)
2.Bash on Ubuntu on Windows上で、リナックス版のAnacondaをインストール。
3.Bash on Ubuntu on Windows上でIPythonと入力すると、IPythonが起動され、IPython上で!前置きによるUNIXコマンドが使えるようになる。
ちなみに、これらの手順のヒント(?)は以下のリンクに記載があり。
Install Anaconda on Windows Subsystem for Linux (WSL) · GitHub