JupyterLabで「Unexpected error while saving file」や「No space left on device」などのエラーが発生した時の対処方法
課題
GCP の AI Platform で JupyterLabを使っていた際に「Unexpected error while saving file」や「No space left on device」などのポップアップが表示され、JupyterLab上での操作ができなくなりました。
解決方法
原因はメモリかハード容量が足りなくなっているためです。私の場合はハード容量に空きをつくることで解決しました。注意点としては、ターミナルでファイルを削除しても、削除したファイルは隠しフォルダ(.local)の中のTrashフォルダに移動されるだけなので、ハード容量の空きは増えません。そのため、ハードの空き容量を増やすには、Trash内のファイルも削除する必要があります。やり方は以下の通りです。
1.ハード容量の空きが無い状態になっているかを確認します。
(base) jupyter@XXXX:~$ df
2.ターミナルを使って不要なファイルを削除します。
(base) jupyter@XXXX:~$ rm <不要なファイル>
3.ファイルを削除後も、ハード容量の空きが無い状態が続いていることを確認します。
(base) jupyter@XXXX:~$ df
4.どこのディレクトリで容量を消費しているのかを確認します。
(base) jupyter@XXXX:~$ du -hs .[^.]*
5.先ほど削除したデータが保存されているTrashまで移動します。
(base) jupyter@XXXX:~$ cd .local/share/Trash/files
6.Trash内のファイルを全て削除すると、JupyterLabで発生していたエラーが消え、JupyterLabの操作が再びできるようになります。
(base) jupyter@XXXX:~/.local/share/Trash/files$ rm *
7.ハード容量の空きが増えていることが確認できればOKです。これでJupyterLabで発生していたエラーが消え、JupyterLabの操作が再びできるようになります。
(base) jupyter@XXXX:~/.local/share/Trash/files$ cd (base) jupyter@XXXX:~$ df
Anaconda環境でgeopandasを使ってshp(シェープ)ファイルを読み込む際に発生するEPSGに関するエラーの対処方法
はじめに
Anaconda環境でgeopandasを使ってshp(シェープ)ファイルを読み込む際に、たまに以下のようなエラーメッセージが出てshpファイルを読み込めない場合があります。
CRSError: Invalid projection: EPSG:4326: (Internal Proj Error: proj_create: SQLite error on SELECT name, type, coordinate_system_auth_name, coordinate_system_code, datum_auth_name, datum_code, area_of_use_auth_name, area_of_use_code, text_definition, deprecated FROM geodetic_crs WHERE auth_name = ? AND code = ?: no such column: area_of_use_auth_name)
geopandasはpyprojパッケージを使って座標系・空間参照のデータベース(EPSG database)を参照しているのですが、その参照先が間違っている場合に発生するようです。
解決方法
import pyproj print(pyproj.datadir.get_data_dir())
の出力が
C:/Users/USERNAME/anaconda3/envs/ENVNAME/lib/site-packages/pyproj/proj_dir/share/proj
の場合、
pyproj.datadir.set_data_dir("C:/Users/USERNAME/anaconda3/envs/ENVNAME/Library/share/proj")
としてpyprojの参照先を現在のAnaconda環境下のLibrary/share/projに変更すると、解決しました。
参考サイト:
CRSError: Invalid projection: EPSG:4326 · Issue #1887 · geopandas/geopandas · GitHub
Sen2Corを使って衛星画像Sentinel-2のL1CをL2Aに変換する方法
はじめに
フリーで使える中解像度の衛星画像データとして、Sentinel-2(分解能10m~)があります。人工衛星Sentinel-2は2015年6月に打ち上げられ、打ち上げ当初からL1Cというタイプの衛星画像が公開されています。L1Cタイプの衛星画像とは、簡単に言うと生データのようなものです。
一方で、2017年3月頃から大気の影響を補正したL2Aタイプの衛星画像の提供も順次開始されています。ヨーロッパ地域から始まり、日本の範囲については2018年12月頃以降のL2Aタイプの衛星画像が提供されています。逆に言うと、例えば日本の2017年分のL2Aタイプの衛星画像は提供されていません。
しかし、自力でL1CをL2Aに変換することは可能です。
L2AはSen2Corというプログラムを使ってL1Cから変換されており、Sen2Corプログラム自体は公式(欧州宇宙機関)が公開しているからです。
この投稿では、Sen2Corを使ってL1Cタイプの衛星画像をL2Aタイプに変換する方法を説明します。ちなみに、PC環境はWindowsです。
1. Sen2Corのインストール
欧州宇宙機関が提供するSen2Corのダウンロードサイトに進み、Sen2Cor_v2.8を選択します。

ページの下の方に進むと、インストーラーの説明があるので、PCのOSに合ったインストーラーを任意の場所にダウンロードします。zipファイルを解凍しておきます。

zipファイルを解凍しておきます。

↓公式サイトのSen2Corインストーラーです。ここからもダウンロードできます。
Windows用:http://step.esa.int/thirdparties/sen2cor/2.8.0/Sen2Cor-02.08.00-win64.zip
Linux用:http://step.esa.int/thirdparties/sen2cor/2.8.0/Sen2Cor-02.08.00-Linux64.run
Mac用:http://step.esa.int/thirdparties/sen2cor/2.8.0/Sen2Cor-02.08.00-Darwin64.run
2. Sentinel-2 L1Cの衛星画像を取得する(※画像を取得済みの場合➝3.へ)
Sentinel-2の衛星画像を提供しているサイトは色々ありますが、今回はCopernicus Open Access Hubから取得します。まずサイトにアクセスし、Open Hubを選択します。

Open Hubに進んだら、右上からアカウントを作成します。

アカウントを作成できたら、衛星画像がほしい範囲を選択します。

左上の三本線を選択します。
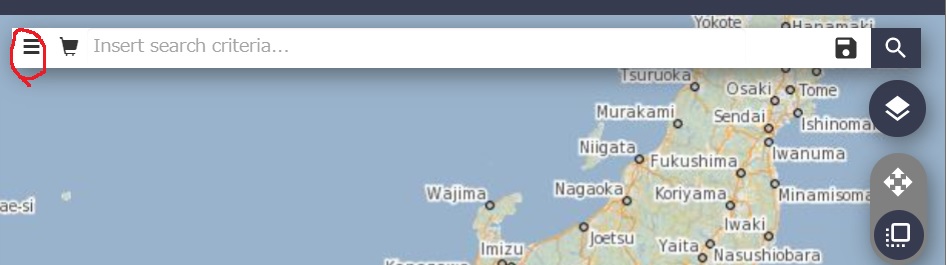
Sensing period(画像の撮影時期)かIngestion period(画像がサイトで公開された時期)の項目に画像の期間を選択します。Mission: Sentinel-2にチェックを入れます。Product TypeはS2MSI1Cを選択します。雲量(Cloud Cover %)を任意の値(例えば10%以下は、[0 TO 10])で入力します。
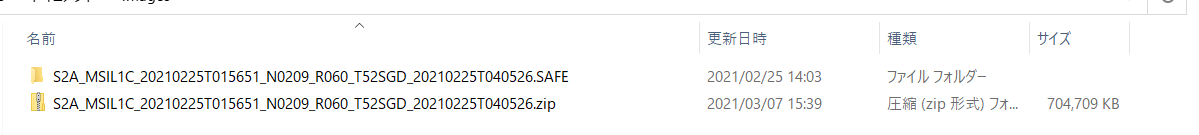
最後に虫眼鏡マークを選択すると、設定した条件に合うSentinel-2のL1Cの衛星画像の一覧が表示されます。この中から必要なデータを選択し、任意の場所にzipファイルをダウンロードし、zipファイルを解凍し、「~.SAFE」というフォルダが生成されるのを確認します。
3. L1CデータをL2Aデータに変換
コマンドプロンプトを開き、Sen2Corのzipファイルを解凍したディレクトリに移動し、ディレクトリ内にL2A_Process.batファイルがあることを確認します。

L2A_Process.batが確認できたら、コマンドラインに
L2A_Process.bat ダウンロードしたL1C画像の.SAFEフォルダのパス
を入力します。
あとは変換作業を待つのみです。パソコンのスペック次第ですが、ノートPC用の第10世代Core-i7だと10分くらいで完了します。以下の画像のような感じで進捗が表示されます。

最後にApplication terminated successfullyの文字が出れば成功です。

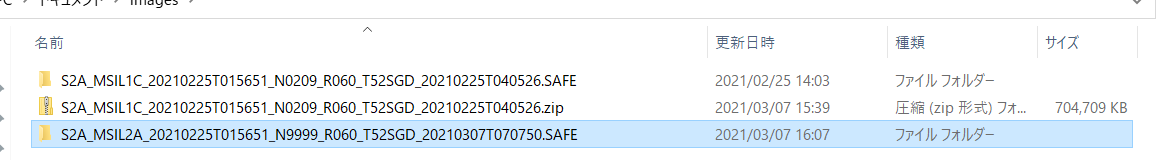
L1Cタイプの画像を保存していたフォルダを確認すると、L2Aタイプの画像が生成されていることが確認できます。
Jupyterのカーネルに設定されたPythonのバージョンを確認する方法
はじめに
Jupyter上で複数の仮想環境を使うために、以下のサイト等を参考にして複数のバージョンのPythonを pyenvで作成した。
・Jupyterで複数カーネルを簡単に選択するための設定 - Qiita
・Python:venv + pyenvでの環境構築 - 無粋な日々に
そして、Jupyter上で各カーネルのPythonのバージョンを
!python --version
で確認したら、ホストのPythonのバージョンが表示されて少し混乱したので、その時の備忘録です。
GPUをWSL2とDockerで使えるようにする方法
はじめに
Dockerの初学者です。Dockerを使い始めるにあたり、WSL2とDockerでGPUを使えるようするために環境の構築を行ったのですが、エラーが連発したので、その記録と解決方法を紹介します。
WSL2上でdockerを使ってGPUを使用する方法は、NVIDIA公式のCUDA on WSL :: CUDA Toolkit Documentationに詳しく説明されています。日本語だとついにWSL2+docker+GPUを動かせるようになったらしいので試してみる - Qiitaが分かりやすいです。私のPCの環境はWindows10 Homeです。
エラー内容①
Docker Desktop for Windowsをインストールして、WSL2と連携したはずなのにWSL2上で以下のコマンドを入力してもDockerが反応しなかった。
$ sudo service docker stop $ sudo service docker start
エラー内容②
WSL2上でGPUが認識されているか確認するためにNVIDIA公式に記載の以下のコードを入力しても、
$ docker run --gpus all nvcr.io/nvidia/k8s/cuda-sample:nbody nbody -gpu -benchmark
以下のようなエラーが発生した。
docker: Error response from daemon: could not select device driver "" with capabilities: [[gpu]]
解決方法
1. PCにDocker Desktop for Windowsがインストール済みの場合は、「アプリと機能」からDocker Desktopをアンインストールする。
2.NVIDIA公式の方法に従って作業する。この時、以下のコードをところで、「既にdockerがマシンに入っているからトラブル起きる可能性あるよ(意訳)」という警告文が発生し、それでも進めようとすると、「WSL2環境があるときにはDocker Desktop for Windowsを使いましょう。(意訳)」と警告文が出るけど無視して進める。
$ curl https://get.docker.com | sh
3.引き続きNVIDIA公式の方法に従って作業し、
$ docker run --gpus all nvcr.io/nvidia/k8s/cuda-sample:nbody nbody -gpu -benchmark
の箇所で、以下のエラーが発生した場合は、
docker: Error response from daemon: cgroups: cannot find cgroup mount destination: unknown. ERRO[0000] error waiting for container: context canceled
以下のようにディレクトを作成してマウントする。
$ sudo mkdir /sys/fs/cgroup/systemd $ sudo mount -t cgroup -o none,name=systemd cgroup /sys/fs/cgroup/systemd
すると、
$ docker run --gpus all nvcr.io/nvidia/k8s/cuda-sample:nbody nbody -gpu -benchmark
が正常に動作します。以上です。
【解決方法の参考サイト】
cuda - GPU/Docker not working Ubuntu 20.04 in the WSL 2 of Windows - Ask Ubuntu
https://github.com/docker/for-linux/issues/219#issuecomment-375160449
Djangoで作ったWebアプリをHerokuでデプロイするとmediaが使えなかった。
課題
Djangoで作ったWebアプリをHerokuで公開したら、DEBUG = True の時はmediaからファイルがダウンロードができたけど、本番環境のDEBUG = False にするとmediaからファイルがダウンロードができなくなった。
原因
PaaS(Herokuなど)ではDjangoのmediaは使えない(※使うためには工夫が必要)。
(参照サイト:Djangoで静的ファイルとうまくやる)
解決策
mediaを使う代わりにドロップボックスにファイルをアップロードして共有URLをHTMLにハイパーリンクとして埋め込む。ちなみに、ドロップボックスの共有URLの末尾をdl=0からdl=1に変更すると、強制ダウンロードにすることができる。
<a href="https://www.dropbox.com/<共有ファイル毎のURL>?dl=1">samplefile.pdf</a>
ポイント
対象のWebアプリでのmediaの用途が、adminが上げたファイルを利用者がダウンロードするためにmediaを使っていただけなので、上記の解決策が楽だった。
Webアプリ
DjangoとHerokuを使ってWebアプリを作りました。
→Google Earth上に風車をつくるWebアプリ